许多蛋白质形状来自科学完全未知的生物体。
Facebook 和 Instagram 的母公司 Meta 的科学家们使用人工智能 (AI) 语言模型来预测属于病毒、细菌和其他微生物的 6 亿多种蛋白质的未知结构。
这个名为 ESMFold 的程序使用了一个最初设计用于解码人类语言的模型,以准确预测决定其 3D 结构的蛋白质所经历的曲折。这些预测被编译到开源 ESM 宏基因组图谱中,可用于帮助开发新药,表征未知微生物功能,并追踪远缘物种之间的进化联系。
ESMFold 并不是第一个进行蛋白质预测的程序。 2022 年,谷歌旗下的公司 DeepMind 宣布其蛋白质预测程序 AlphaFold 已经破译了科学界已知的大约 2 亿种蛋白质的形状。 Meta 说,ESMFold 不如 AlphaFold 准确,但比 DeepMind 的程序快 60 倍。结果尚未经过同行评审。
“ESM 宏基因组图谱将使科学家能够在数亿蛋白质的规模上搜索和分析宏基因组蛋白质的结构,”Meta 研究小组在一篇博客文章中写道,该文章伴随着该论文发布到预印本数据库 bioRxiv。 “这可以帮助研究人员识别以前未被表征的结构,寻找遥远的进化关系,并发现可用于医学和其他应用的新蛋白质。”
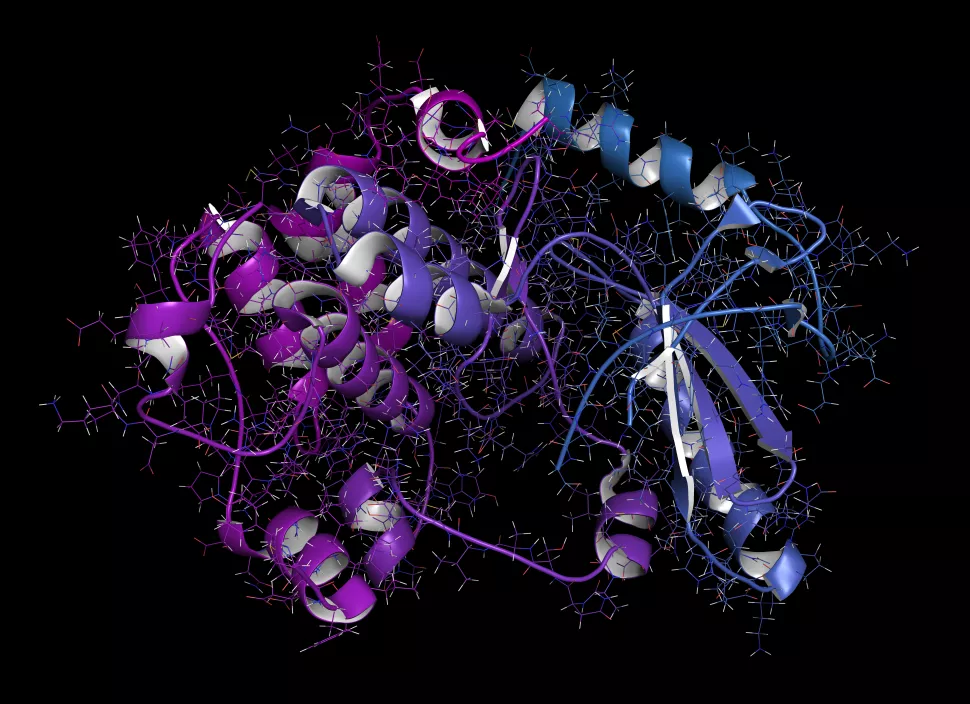
蛋白质是所有生物的组成部分,由长而曲折的氨基酸链组成——这些微小的分子单元以无数种组合形式结合在一起形成蛋白质的 3D 形状。
了解蛋白质的形状是了解其功能的最佳方式,但不同序列中的相同氨基酸组合可以通过多种方式形成。尽管蛋白质一旦被生产出来就可以快速可靠地形成某些形状,但可能的配置数量大约为 10^300。确定蛋白质结构的黄金标准方法是使用 X 射线晶体学——观察高能光束如何在蛋白质周围衍射——但这是一种艰苦的方法,可能需要数月或数年才能产生结果,而且它不起作用适用于所有蛋白质类型。经过数十年的工作,已经通过 X 射线晶体学破译了超过 100,000 种蛋白质结构。
为了找到解决这个问题的方法,Meta 研究人员转向了一种复杂的计算机模型,该模型旨在解码和预测人类语言,并将该模型应用于蛋白质序列的语言。
研究人员写道:“使用一种称为掩码语言建模的自我监督学习形式,我们在数百万个天然蛋白质的序列上训练了一个语言模型。” “通过这种方法,模型必须正确填写一段文本中的空白,例如“To __ or not to __,即 ________。”我们训练了一个语言模型来填补蛋白质序列中的空白,就像跨越数百万种不同蛋白质的“GL_KKE_AHY_G”一样。我们发现有关蛋白质结构和功能的信息来自这种训练。”
为了测试他们的模型,科学家们求助于一个宏基因组 DNA 数据库(之所以如此命名,是因为它已从环境或临床来源大量测序),取自土壤、海水以及人类肠道和皮肤等不同地方。通过将 DNA 数据输入 ESMFold 程序,研究人员在短短两周内预测了超过 6.17 亿种蛋白质的结构。
这比 AlphaFold 宣布的四个月前破译的数量多 4 亿多,当时它声称已经推断出几乎所有已知蛋白质的蛋白质结构。这意味着其中许多蛋白质以前从未见过,可能是因为它们来自未知生物。根据该模型,ESMFold 的超过 2 亿个蛋白质预测被认为是高质量的,这意味着该程序能够以低至原子水平的精度预测形状。
研究人员希望将这个程序用于更多以蛋白质为重点的工作。 “为了进一步扩展这项工作,我们正在研究如何使用语言模型来设计新的蛋白质,并有助于解决健康、疾病和环境方面的挑战,”Meta 写道。